Die Entstehung der zellulären Informationsübertragung stellt die Wissenschaft bis heute vor ein großes Rätsel. Die auf unseren Genen festgehaltene Information, ähnlich den Daten auf einer Festplatte, liegt in codierter Form vor und muss interpretiert werden. Im Anschluss wird diese Information als eine Art Blaupause genutzt, um Proteine zu erzeugen, die wiederum die verschiedensten Aufgaben in der Zelle übernehmen. Doch wie ist dieses ausgeklügelte System der Informationsweiterleitung zustande gekommen? Wie haben sich der genetische Code und die Proteine, die ihn übersetzen, im Laufe der Zeit entwickelt, um das Leben wie wir es kennen zu ermöglichen?
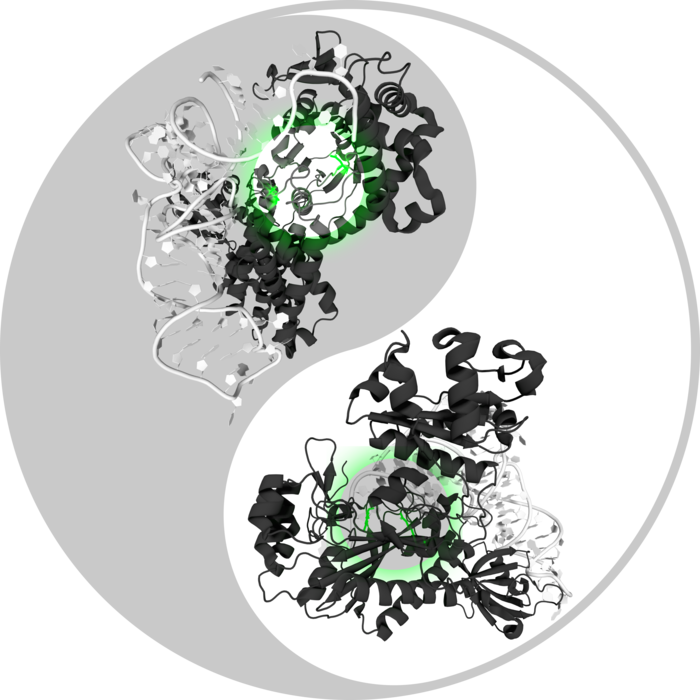
An der Schnittstelle zwischen genetischer Information und deren Interpretation steht eine besondere Klasse von Enzymen, die sog. Aminoacyl-tRNA Synthetasen. Die Entstehung des genetischen Codes ist eng mit der Geschichte der Aminoacyl-tRNA Synthetasen verwoben. Diese Enzyme waren unter den ersten zellulären Maschinen, die die Evolution hervorgebracht hat. Da Aminoacyl-tRNA Synthetasen den genetischen Code manifestieren und selbst in diesem codiert sind, ist die Frage ihrer Entstehung ein Henne-Ei-Problem.
Die von Prof. Labudde geleitete Forschungsgruppe für Bioinformatik bigM wurde durch eine Kooperation mit Prof. Peter Wills auf dieses spannende Thema aufmerksam als er im November 2016 seine Forschungsarbeit in Mittweida vorstellte. Daraufhin wurden diese Proteine im Rahmen einer Kooperation der bigM und der TU Dresden mit bioinformatischen Methoden untersucht.
Die Forschungsergebnisse dieser Arbeit wurden nun im renommierten Fachjournal PLOS Computational Biology veröffentlicht. Der Artikel trägt den Titel “Backbone Brackets and Arginine Tweezers delineate Class I and Class II aminoacyl tRNA synthetases”. Als besondere Auszeichnung wurde der Beitrag auf der Titelseite der April-Ausgabe des Journals abgedruckt.
Die Forscher konnten zum ersten Mal zeigen, dass Aminoacyl-tRNA Synthetasen zwei komplementäre molekulare Bausteine enthalten, die seit Anbeginn der Entstehung des genetischen Codes vorhanden sind. Die Ergebnisse komplementieren bestehende Studien und weisen darauf hin, dass Aminoacyl-tRNA Synthetasen von einem gemeinsamen “Ur-Gen” abstammen, welches alle heutigen Organismen miteinander verbindet.
Text: Florian Kaiser