Fokus Forschung: Entwicklung eines genetischen Biomonitoring Protokolls zur Untersuchung von Bestäuber-Pflanze-Netzwerken
Fokus Forschung: Entwicklung eines genetischen Biomonitoring Protokolls zur Untersuchung von Bestäuber-Pflanze-Netzwerken
Forscher Nachwuchs | Lisa Prudnikow entwickelt Biodiversitäts-Test für Bienen
Unsere Zivilisation muss sich mit einem enormen globalen Rückgang der Artenvielfalt auseinandersetzen. Besonders Insekten sind bedroht [1]. Ihre Dienste wie zum Beispiel die Blütenbestäubung sind aber absolut unverzichtbar für ein stabiles Ökosystem. Außerdem sorgen sie für die genetische Durchmischung von Wild- und Nutzpflanzen [2]. Doch durch die Intensivierung der Landwirtschaft, werden Agrarsysteme strukturell monotonisiert. Das Nahrungsangebot für Bestäuber in Form von Blütenpollen und Nektar vereinfacht sich dementsprechend ebenfalls [3]. Aber gerade die Diversität von Landschaften sorgt für ein vielfältiges Pollenangebot, das sich positiv auf das Immunsystem von Bestäubern auswirkt. Eine einseitige Pollenernährung führt zu Nährstoffmangel und gegebenenfalls sogar zum Verlust eines kompletten Bienenvolkes durch Mangelernährung und Schwächung [4].
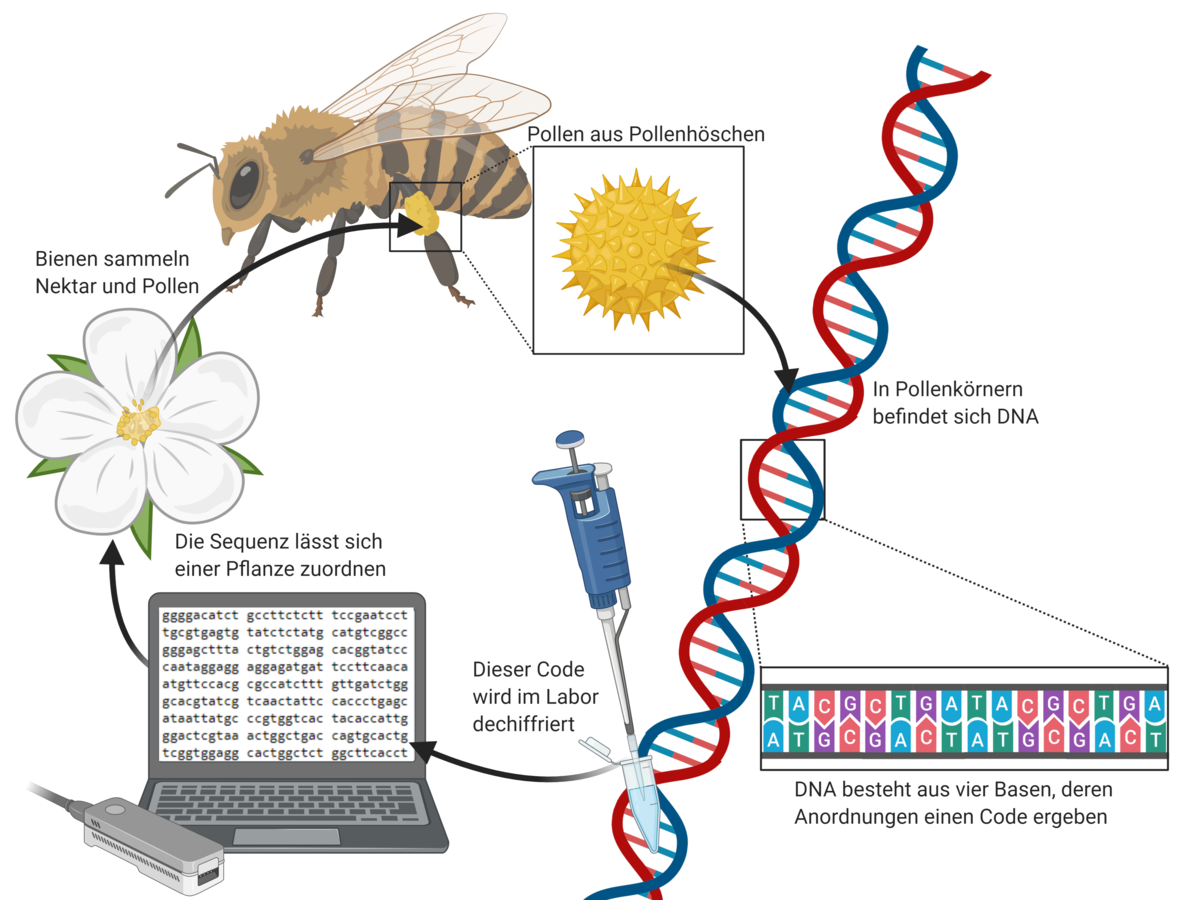
Um sinnvolle Managementstrategien und Konzepte zur Erhaltung oder Wiederherstellung der biologischen Vielfalt zu etablieren, müssen Informationen über das Vorhandensein von Flora und Fauna in verschiedenen Landschaftsszenarien gesammelt und ausgewertet werden. Solche Methoden werden als Biomonitoring zusammengefasst [1]. Informationen über Bestäuber-Pflanze-Netzwerke sind von Nutzen, da sie dazu beitragen können, Agrarsysteme im Sinne der Biodiversität nachhaltig zu verändern [3]. Das heißt, dass das Sammelverhalten von Bestäubern in verschiedenen Habitaten dokumentiert wird [5]. Die Analyse der Erbinformation in durch Bienen eingetragenen Pollenkörnern, kann auf die Herkunftspflanze zurückweisen. Dafür muss die DNA aus den Pollen isoliert werden, damit sie mit einem Sequenzierungsgerät entschlüsselt werden kann [6].
Mit dieser Arbeit soll ein Protokoll entwickelt werden, das die DNA von durch Bestäubern eingetragenen Pollen mit Hilfe der Nanoporensequenzierung entschlüsselt. Dafür müssen Parameter für eine ertragreiche Pollen-DNA-Extraktion, der anschließenden DNA-Sequenzierung sowie einer Pipeline für eine umfassende Datenauswertung festgelegt werden. Der entstehende Methoden-Leitfaden soll eine weitere Grundlage für eine nachhaltige Biodiversitätsforschung und zukünftige ökologische Fragestellungen darstellen.
Um dies zu erreichen, wird in Zusammenarbeit mit der Georg-August-Universität Göttingen und Imkern des Imkerverein Chemnitz 1874 e.V. Pollen, der von Honigbienen (Apis mellifera), Hummeln (Bombus terrestris) und Mauerbienen (Osmia bicornis) gesammelt wird, genutzt. Die Pollenhöschen bzw. die von Mauerbienen eingetragenen Pollenklumpen werden geerntet und für die DNA-Sequenzierung vorbereitet. Da Pollen einen sehr robusten Zelltyp darstellt [7], muss zunächst an einer effektiven Methode zum Zellaufschluss gearbeitet werden. Mit Metall- bzw. Keramik-Beads werden die Pollenproben behandelt und anschließend mit dem DNeasy® Plant Mini Kit (Qiagen) die DNA extrahiert. Die isolierte Pollen-DNA soll anschließend durch Metabarcoding identifiziert werden. Das heißt, dass anhand eines bestimmten Abschnitts (Barcode) auf der DNA ein Organismus ermittelt wird. Die Internal Transcribed Spacer Region (ITS2) ist ein bei Pflanzen häufig verwendeter Barcode [8], der hier eingesetzt werden soll. Zur Sequenzierung wird der von Oxford Nanopore Technologies entwickelte Flongle Sequencer verwendet. Dieser portable Nanoporen-basierte Sequenzierautomat ermöglicht eine schnelle Analyse von DNA-Proben vor Ort und vermeidet den Transport in weit entfernte Labore [1]. Dass DNA entschlüsselt werden kann, wird mit dem Prinzip des Membranpotential erreicht. Mithilfe eines Motorproteins passiert einzelsträngige DNA Tunnelproteine in nm-Größe, die in eine Bilipid-Membran eingebettet sind. Beim Anlegen einer externen einseitigen Spannung kann der Ionenstrom gemessen werden, der charakteristischerweise durch die eingegebene DNA blockiert wird [9]. Mit dieser Technologie soll ein genetischer Biodiversitäts-Test entwickelt werden, um die florale Biodiversität eines Standorts und das Sammelverhalten von Bestäubern zu bewerten. Die entstandenen Sequenzdaten werden bioinformatisch prozessiert und mit einem Datenbankabgleich anhand der ITS2-Region die Herkunftspflanzen identifiziert. Die entsprechende ITS2-Sequenz-Datenbank wurde dafür mit einer Liste von Pflanzen, die in Sachsen bzw. Deutschland vorkommen [10, 11, 12, 13], erstellt. Mit den erlangten Daten über die Herkunft der Pollen, die die Bestäuber sammeln, können danach umfassende Biodiversitäts-Analysen durchgeführt werden (Shannon- und Simpsonindex, Food webs usw.). Da Pollen von den Bestäubern an mehreren Standorten geerntet wird, soll neben der Etablierung des Protokolls auch Fragestellungen geklärt werden: 1. Von welchen Pflanzen sammeln die drei Bestäuberarten die meisten Pollen? 2. Teilen Sie in ihrem Sammelverhalten maßgeblich bestimmte Pollenspezies und zu welchem Anteil? 3. Können Unterschiede in der Pollenernährung über den Zeitraum der Studie (Mai bis August) festgestellt werden? Antworten auf diese Fragen sollen zu einem funktionellen Verständnis von Bestäuber-Pflanze-Netzwerken beitragen.
Im Rahmen ihrer Masterarbeit beschäftigt sich Lisa Prudnikow gemeinsam mit Prof. Röbbe Wünschiers und Robert Leidenfrost mit der genetischen Pollenanalyse. Lisa Prudnikow absolvierte bereits den Bachelorstudiengang „Biotechnologie“ in Mittweida und schloss mit dem in 2019 neu eingeführten Master “Genomische Biotechnologie” an ihre Ausbildung an.
Text:. Lisa Prudnikow, Prof. Röbbe Wünschiers, Robert Leidenfrost (Hochschule Mittweida); Prof. Catrin Westphal (Georg-August-Universität Göttingen)
Foto: Hochschule Mittweida
Abbildung: Lisa Prudnikow


Verwendete Literatur
[1] Krehenwinkel, H.; Pomerantz, A. & Prost, S. (2019) Genetic Biomonitoring and Biodiversity Assessment Using Portable Sequencing Technologies: Current Uses and Future Directions. Genes. 10(11):858. DOI: 10.3390/genes10110858
[2] Bender, S.; Chemnitz, C. & Klein, A.-M. et al. (2020) Insektenatlas – Daten und Fakten über Nütz- und Schädlinge in der Landwirtschaft. Heinrich-Böll-Stifutung. 2: Auflage. ISBN: 978-386928-215-2
[3] Bänsch, S. (2019) Managing strawberry pollination with wild bees and honey bees: Facilitation or competition by mass-flowering resources? 136 pages. Göttingen, Georg-August-Universität Göttingen, department of agroecology, Dissertation,
[4] Di Pasquale, Garance; Salignon, M. & Le Conte, Y. et al. (2013) Influence of Pollen Nutrition on Honey Bee Health: Do Pollen Quality and Diversity Matter? PLOS ONE. 8(8): e72016. DOI: 10.1371/journal.pone.0072016
[5] Kamo, T.; Kusumoto, Y. & Tokuoka, Y. (2018) A DNA barcoding method for identifying and quantifying the composition of pollen species collected by Europoean Honeybees , Apis mellifera (Hymenoptera: Apidae). Applied Entomology and Zoology. 53:353-361. DOI: 10.1007/s13355-018-0565-9
[6] Ruppert, K., Kline, R. & Rahman, S. (2019) Past, Present, and future perspectives of environmental DNA (eDNA) metabarcoding: A systematic review in methods, monitoring, and applications of global eDNA. Global ecology and conservation. 17:e00547. DOI: 10.1016/j.gecco.2019.e00547
[7] Halbritter, H.; Ulrich, S. & Grímsson, F. et al. (2018) Illustrated Pollen Terminology. Second Edition. Springer Verlag. DOI: 10.1007/978-3-319-71365-6
[8] Yao, H.; Song, J. & Liu, C. et al. (2010) Use of ITS2 Region as the Universal DNA Barcode for Plants and Animals. PLoS one. 5(10):e13102. DOI:10.1371/journal.pone.0013102
[9] van Dijk, E.; Jaszyszyn, Y. & Naquin, D. et al. (2018): The third Revolution in Sequencing Technology. Trends in Genetics. 34(9):666-681. DOI:10.1016/j.tig.2018.05.008
[10] Förster, F. (2015) eukaryota.all.fasta.bz2. Retrieved from: github.com/BioInfWuerzburg/ITS2database_update_2015/blob/master/data/2015/eukaryota.all.fasta.bz2.
[11] “Artenreferenzliste Sachsen”. Unknown author. Retrieved from: www.natur.sachsen.de/artenreferenzliste-sachsen-21920.html
[12] „Gebietseigene Pflanzen“. Artenlisten Sachsen. Unknown author. Retrieved from: www.natur.sachsen.de/gebietseigene-pflanzen-20826.html
[13] Ludwig, G. & Schnittler, M. (1996) Schriftenreihe für Vegetationskunde 28