Fokus Forschung: Vom Plasmid-Design zur in vitro transkribierten RNA: Etablierung einer kompletten RNA-Produktionslinie an der Hochschule Mittweida
Fokus Forschung: Vom Plasmid-Design zur in vitro transkribierten RNA: Etablierung einer kompletten RNA-Produktionslinie an der Hochschule Mittweida
Forscher Nachwuchs | Vanessa Schumann forscht zur Herstellung von RNA-Tertiärkontakten
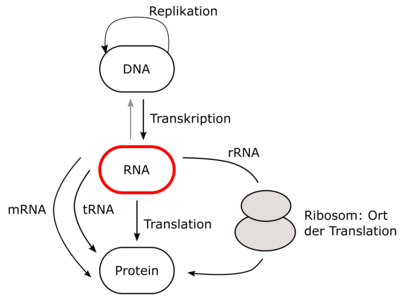
Die Ribonukleinsäure (RNA) ist eines der wichtigsten Biomoleküle, das wir kennen. RNA ist an den meisten essenziellen Prozessen innerhalb einer Zelle beteiligt und trägt eine katalytische oder regulatorische Funktion. RNA ist außerdem ein wichtiger Baustein bei der Übersetzung der genetischen Information aus der DNA-Sequenz in effektiv verwertbare Informationen für den Proteinaufbau (Abbildung 1) [1].
RNA-Moleküle sind aus Ribonukleotiden aufgebaut. Ein Ribonukleotid besteht immer aus einem Zuckermolekül, der Ribose, einer Phosphatgruppe und einer der vier Basen Adenin, Guanin, Cytosin oder Uracil. Diese Nukleosidmonophosphate sind durch Phosphorsäurediesterbindungen miteinander verbunden. Im Gegensatz zur DNA, die als Doppelhelix vorliegt, ist die RNA meist einzelsträngig [2]. Die reine Abfolge dieser Nukleotidsequenz wird als Primärstruktur bezeichnet. Damit die RNA aber ihre Funktionen erfüllen kann, muss sie sich oft erst in ihre spezifische dreidimensionale Form falten. Während dieses Faltungsprozesses entstehen aus der Primärstruktur Motive wie Basen-gepaarte Heizes, Haarnadelschleifen oder Verzweigungen, welche die Sekundärstruktur bilden. Interagieren nun diese Sekundärstrukturelemente miteinander entsteht die dreidimensionale Tertiärstruktur. Sogenannte RNA-Tertiärkontakte wie Kissing Loops und Pseudoknoten sind essenziell für die Stabilisierung der dreidimensionalen Faltung der RNA. Die Faltungsdynamik solcher Tertiärkontakte kann Einblicke in RNA-RNA-Wechselwirkungen geben und erlaubt es, die Kinetik und Thermodynamik dieser Wechselwirkungen näher zu charakterisieren [3].
Zusammen mit Prof. Richard Börner forsche ich an einer Produktionslinie, um einen solchen RNA-Tertiärkontakt herzustellen. Dieser besondere Tertiärkontakt beeinflusst die Faltung der ribosomalen RNA (rRNA) und soll in weiteren Arbeiten näher untersucht werden [4].
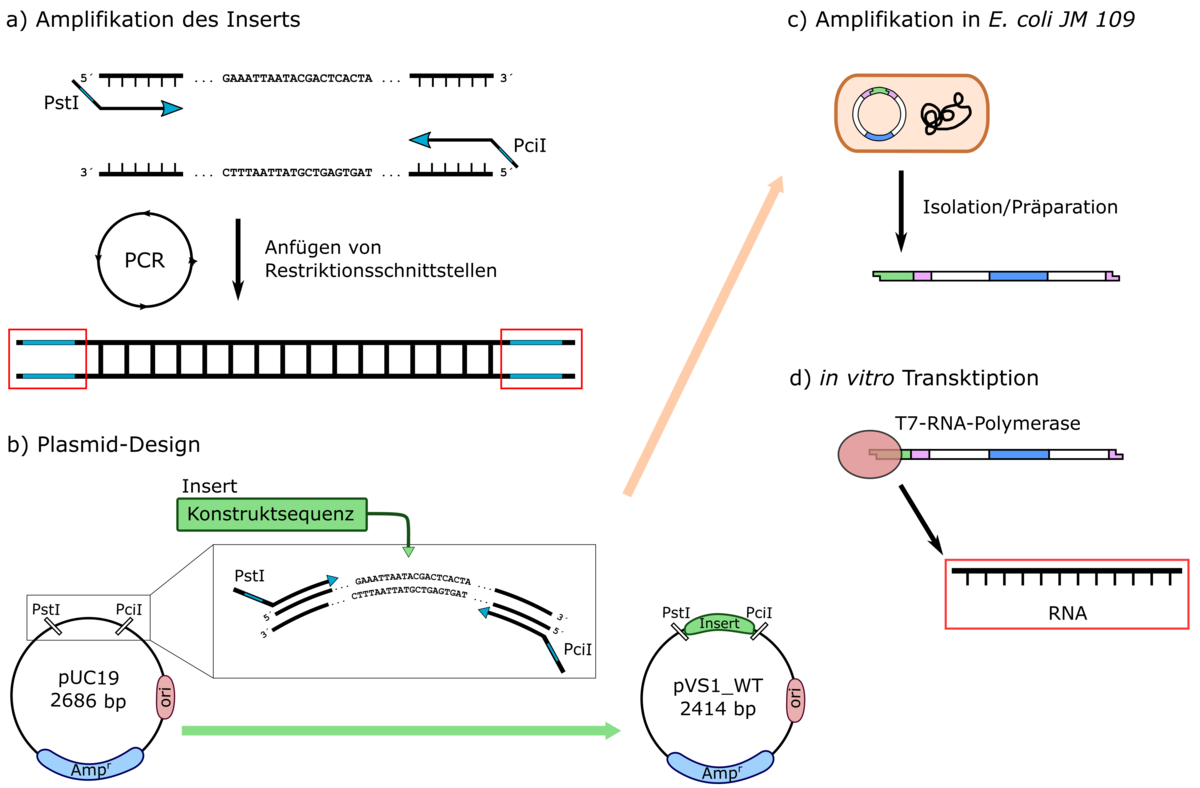
Zur Etablierung der RNA-Produktionslinie werden hauptsächlich Methoden des molecular cloning angewandt. Das heißt: Ein sogenanntes „Insert“ wird in ein Vektorplasmid eingefügt. In unserem Fall ist das Insert die RNA-Sequenz. Um das Insert in das Vektorplasmid einfügen zu können, muss es Schnittstellen für Restriktionsenzyme erhalten. Dies geschieht während einer Polymerase-Kettenreaktion (PCR). Dazu wurden spezifische Primer entworfen, welche die entsprechenden Erkennungssequenzen der Restriktionsenzyme enthalten. Wurden die Schnittstellen an das Insert angefügt, kann es durch Restriktionsverdau und Ligation in das Vektorplasmid eingefügt werden. Anschließend soll das neu erhaltene Plasmid in E. coli kloniert werden, um ausreichende Mengen des Plasmids amplifizieren zu können. Durch nachfolgende Plasmid-Reinigungsschritte soll ein linearisiertes Plasmid für die in vitro-Transkription der RNA, mittels T7-RNA-Polymerase, vorliegen [5,6]. Die komplette Produktionslinie ist in Abbildung 2 dargestellt.
Ich bin Vanessa Schumann und studiere seit 2016 an der Hochschule Mittweida. Nach meinem Bachelor in Biotechnologie entschied ich mich, ebenfalls den Master hier an der HSMW, im Studiengang Genomische Biotechnologie mit der Vertiefung Molekularbiologie, zu absolvieren. Während dieser Zeit lernte ich Prof. Börner und seine Forschungsgruppe kennen und bekam die Möglichkeit, sowohl mein Forschungsmodul, als auch die Masterarbeit im in der Forschungsgruppe durchzuführen. Neben dem Studium bin ich in der Studienberatung unserer Hochschule tätig und ehrenamtliches Mitglied in der DRK-Wasserwacht. Mein Dank gilt Prof. Richard Börner für das spannende Forschungsthema sowie dem Laserinstitut Hochschule Mittweida und der Hochschule Mittweida für die finanzielle Unterstützung bei der Durchführung meiner Forschungsarbeit. Weiterhin danke ich allen, die mich bei meiner Arbeit unterstützt haben, insbesondere Prof. Röbbe Wünschiers für die Möglichkeit in der „Biobox“ der Hochschule Mittweida arbeiten zu dürfen sowie Dr. Susann Zelger-Paulus von der Universität Zürich und Sandra Feik für zahlreiche wissenschaftliche Diskussionen.
Literaturnachweis
[1] Crick, F.: „Central dogma of molecular biology“, In: Nature 227 (1970)
[2] J. Arnemann. Lexikon der Medizinischen Laboratoriumsdiagnostik. Ed. by A. M. Gressner and T. Arndt. Springer-Verlag Berlin Heidelberg, 2. Auflage, 2009. ISBN: 978-3-642-34635-4.
[3] Tinoco, I; Bustamate, C.: „How RNA folds“, In: Journal of Molecular Biology 293.2 (1999)
[4] Stefan Gerhardy, Michaela Oplova, Richard Börner, Ludovic Gillet, Rob van Nues, Ahmed Moursy, Alain Scaiola, Alexander Leitner, Daniel Böhringer, Martin Altvater, Janusz Petkowski, Roland K.O. Sigel, Nenad Ban, Frederic Allain, Sander Grannemann, Ruedi Aebersold, Vikram Govind Panse: „Puf6 ushers correct ribosomal RNA folding during 60S assembly” (2021) under review
[5] Gallo, S.; Furler, M.; Sigel, R. K. O.: „In vitro Transcription and Purification of RNAs of Different Size“, In: CHIMIA 59.11 (2005) [6] Sambrook, J.; Russell, D. W.: „Molecular Cloning – A Laboratory Manual“, Third Edition, Vol. 1,2; Cold Spring Harbor Laboratory Press (2001)
Text, Foto und Abbildungen: Vanessa Schumann